Die Einzelmolekül-Lokalisierungsmikroskopie ist eine leistungsstarke Methode, um zelluläre Strukturen mit Nanometerauflösung abzubilden. Wir haben DECODE entwickelt, einen auf Deep Learning basierenden Analysealgorithmus, der diese Technik schneller und noch präziser macht.
Mit der superauflösenden Mikroskopie können Wissenschaftlerinnen und Wissenschaftler bisher unentdeckte zelluläre Welten erkunden. So können sie zum Beispiel die subzelluläre Architektur und molekulare Interaktionen im Nanobereich untersuchen. Diese Methoden revolutionierten die Lichtmikroskopie und brachten ihren Erfindern 2014 den Nobelpreis für Chemie ein.
Um die Bedeutung dieser Technologie besser zu verstehen, ist es hilfreich, sich zunächst die Grenzen der bisherigen Techniken vor zu Augen führen. Bei der Fluoreszenzmikroskopie wird das Licht gemessen, das von sogenannten Fluorophoren ausgesandt wird, die an die zu erforschenden biologischen Strukturen gebunden sind. Die Auflösung, die man auf diese Weise erreichen kann, ist – wie auch bei anderen Formen der Lichtmikroskopie – durch die physikalischen Eigenschaften des Lichts selbst begrenzt. Wenn wir winzige Objekte betrachten, zum Beispiel ein fluoreszenzmarkiertes Molekül, sehen wir einen hellen Fleck, der viel größer ist als das Molekül selbst. Wenn zwei solche Lichtquellen zu nah beieinander liegen, verschwimmen ihre Bilder ineinander und man kann sie nicht mehr voneinander unterscheiden.
Methoden der superauflösenden Mikroskopie überwinden diese Grenze. Eine davon ist die sogenannte Einzelmolekül-Lokalisationsmikroskopie, bei der ein einfacher Trick angewandt wird: Anstatt alle Fluorophore gleichzeitig zu aktivieren, werden zu jedem Zeitpunkt nur einige wenige zufällig aktiviert. Das führt zu einer Reihe von Bildern mit voneinander getrennten Flecken, die mit hoher Präzision lokalisiert werden können. Anschließend werden die Lokalisierungen aus vielen solcher Bilder miteinander kombiniert, um ein hochaufgelöstes Bild zu rekonstruieren.
Diese Methode hat jedoch einen großen Nachteil: Um eine hohe Lokalisierungsgenauigkeit zu erreichen, müssen die Flecken in der Regel gut voneinander getrennt sein. Daher musste man bisher sicherstellen, dass in jedem Bild nur wenige Flecken sichtbar waren, was wiederum zu langen Aufnahmezeiten führte.
Deep Learning ermöglicht eine präzise Lokalisierung unter schwierigen Bedingungen
In einer Zusammenarbeit zwischen den Gruppen von Jakob Macke (Uni Tübingen), Jonas Ries (EMBL Heidelberg) und Srinivas Turaga (Janelia Research Campus, Virginia, USA), unter Federführung von Artur Speiser und Lucas-Raphael Müller, haben wir den Algorithmus DECODE entwickelt, der diese Einschränkung überwindet. Er erkennt und lokalisiert Fluorophore auch dann, wenn sie dicht beieinander liegen, was eine bis zu zehnmal höhere Bildgebungsgeschwindigkeit ohne nennenswerten Verlust an Auflösung ermöglicht. Unsere Ergebnisse wurden in Nature Methods veröffentlicht.
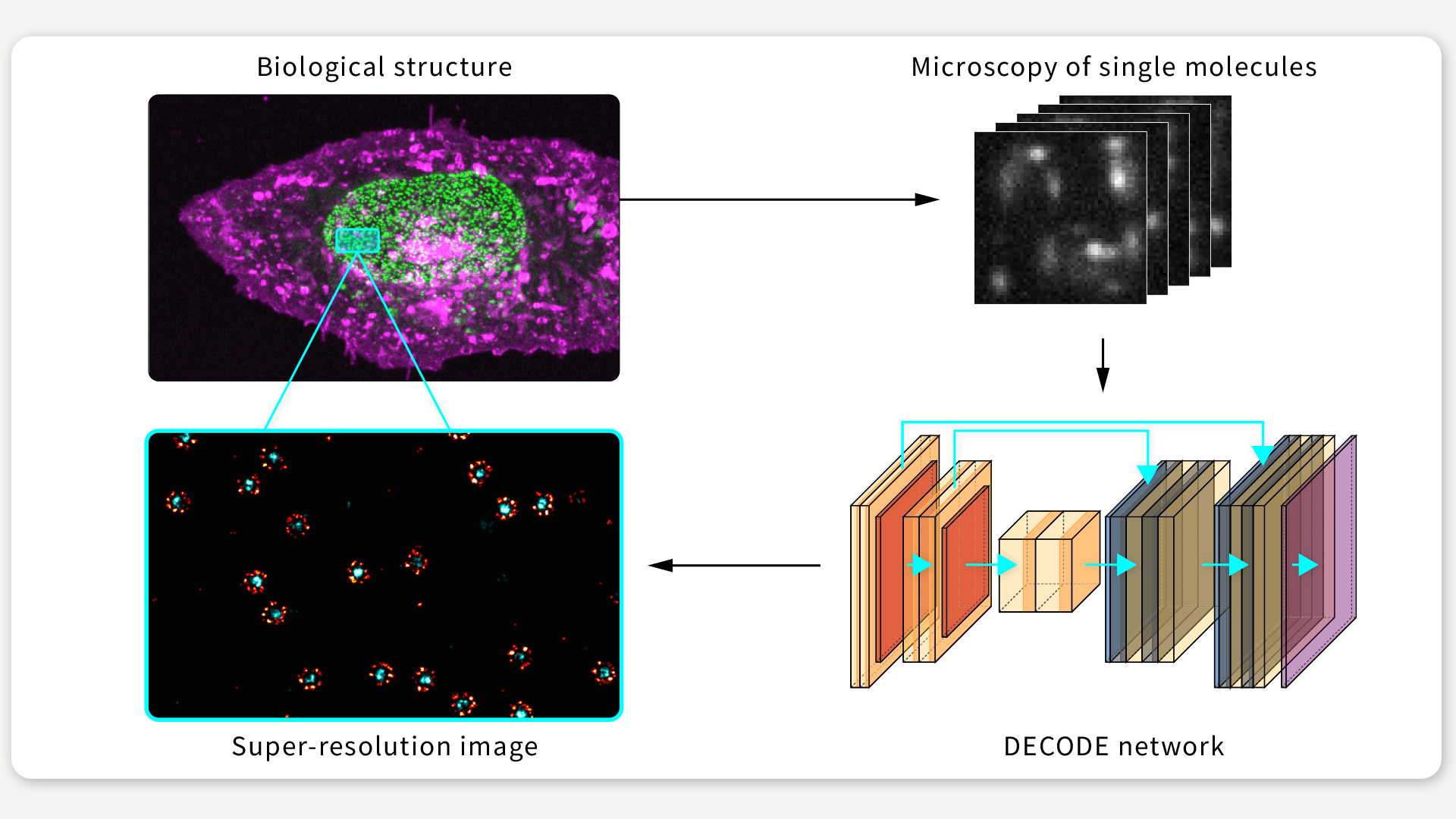
ABBILDUNG 1 / DECODE ist ein neuer Algorithmus, der die Leistung der Einzelmolekül-Lokalisierungsmikroskopie erhöht. Abbildung: Jonas Ries
Unser Algorithmus basiert auf Deep Learning: Wir verwenden ein künstliches neuronales Netz, das Kamerabilder als Eingabe verwendet und Vorhersagen über die Positionen der Fluorophore erstellt (siehe Abbildung 1). Damit das funktioniert, mussten wir mehrere Herausforderungen lösen.
Um neuronale Netze zu trainieren, braucht man Paare aus Eingaben und gewünschten Ausgaben des Netzes, in unserem Fall die Positionen der Fluorophore. In unserer Anwendung wissen wir aber nicht, wo genau sich die Fluorophore befinden. Daher haben wir eine Methode verwendet, die in letzter Zeit für das Training neuronaler Netze in solchen Situationen populär geworden ist und die unsere Gruppe auch in anderen Zusammenhängen erfolgreich eingesetzt hat. Anstatt das Netz mit echten Bildern zu trainieren, haben wir synthetische Daten benutzt, die wir durch numerische Simulationen erzeugt haben. Indem wir unser Wissen über das Mikroskop, die Kamera und die Fluoreszenzmuster der Moleküle mit einbezogen haben, erreichten wir eine große Ähnlichkeit zwischen realen Daten und Simulationen.
Netze, die für die Lokalisierung von Fluorophoren auf simulierten Daten trainiert wurden, sind somit auch in der Lage, dies auf echten Bildern zu tun!
DECODE ermöglicht zehnmal höhere Bildgebungsgeschwindigkeiten
Darüber hinaus haben wir eine Netzwerkarchitektur und einen Trainingsansatz entwickelt, der es erlaubt Fluorophore mit einer Genauigkeit zu lokalisieren, die nah an der theoretischen Grenze liegt. Gleichzeitig liefert das trainierte Netzwerk Unsicherheitsschätzungen, d. h. es gibt Aufschluss darüber, wie gut die einzelnen Lokalisierungen sind. Indem wir schlechte Lokalisierungen beseitigen, können wir die Bildauflösung weiter verbessern.
Wie Abbildung 2 zeigt, erreicht DECODE selbst in solchen Datensätzen eine hohe Auflösung, in denen etwa zehnmal mehr Fluorophore pro Bild aktiviert sind, als es bei dieser Mikroskopietechnik üblich ist.
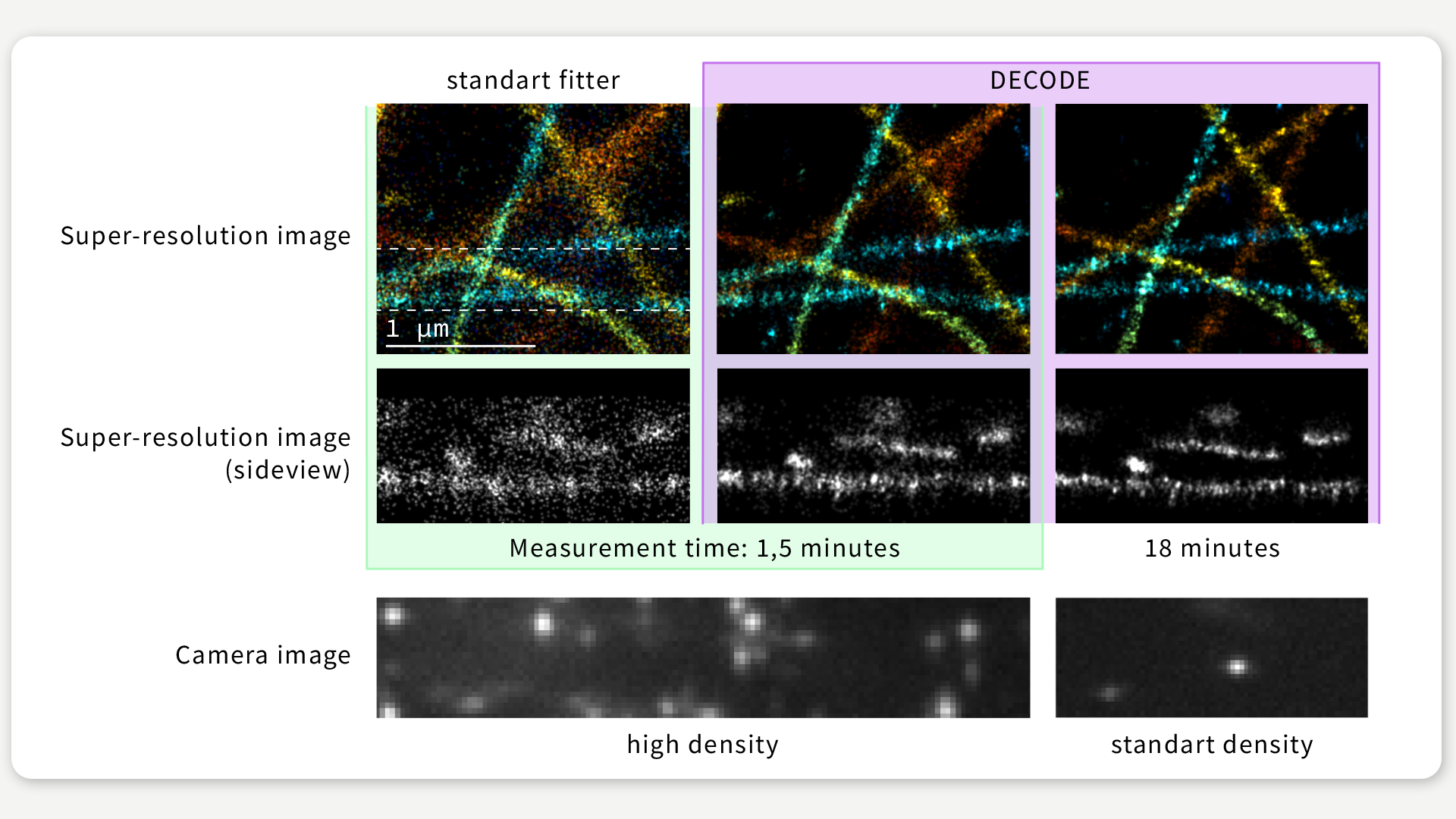
ABBILDUNG 2 / Wir haben dieselbe biologische Struktur unter verschiedenen Bedingungen abgebildet, was zu höheren Aktivierungsdichten und damit zu niedrigeren Abbildungszeiten führte. Selbst bei einer zwölffach höheren Bildgebungsgeschwindigkeit verschlechtert sich die mit DECODE erzielte Auflösung nur geringfügig. Dagegen liefert CSpline, ein anderer beliebter Algorithmus für die Einzelmolekül-Lokalisierungsmikroskopie, eine weit weniger detaillierte Rekonstruktion. Abbildung: Jonas Ries
Wie gut DECODE im Vergleich zu anderen Algorithmen ist, hat ein öffentlicher Wettbewerb gezeigt. Dabei wurden verschiedene Softwares für die Einzelmolekül-Lokalisierungsmikroskopie miteinander verglichen. Verwendet wurden dabei realistische, simulierte Datensätze. Das Ergebnis: DECODE hat alle 39 Algorithmen unter allen Datendichten und experimentellen Bedingungen übertroffen – oft sogar mit deutlichem Abstand. Das bedeutet, dass DECODE immer der am besten geeignete Algorithmus ist, nicht nur bei besonders dichten Daten.
Mit DECODE können viele Forschende ihre Bilder verbessern
Unsere Arbeit zeigt, wie ein neuer Ansatz für ein scheinbar einfaches Problem („wie kann man Flecken in einem Bild präzise lokalisieren?“) zu erheblichen Vorteilen führen kann. Zudem haben wir ein Softwarepaket entwickelt, das den DECODE-Algorithmus implementiert und das online verfügbar ist. Wir gehen davon aus, dass viele Forschende, die die Einzelmolekül-Lokalisierungsmikroskopie nutzen, ihre Bilder verbessern können, indem sie einfach ihre Analysesoftware auf DECODE umstellen. Das funktioniert selbst dann, wenn sie mit ihren bestehenden Datensätzen arbeiten. Insgesamt zeigt DECODE, wie gut sich numerische Simulationen dazu eignen, neuronale Netze zu trainieren. So lassen sich anspruchsvolle Analyseprobleme in den Naturwissenschaften lösen.
Originalpublikation: Speiser A., Muller L.-R. et al. Deep learning enables fast and dense single-molecule localization with high accuracy. Nat. Methods. https://doi.org/10.1038/s41592-021-01236-x (2021).
Text: Artur Speiser
Titelbild: COS-7-Zelle, aufgenommen mit der Einzelmolekül-Lokalisierungsmikroskopie. Rekonstruktion von Artur Speiser et. al., Daten bereitgestellt von Wesley Legant et. al.

Mit maschinellem Lernen Modelle in der Neurowissenschaft identifizieren

Kommentare