Das Gehirn ist zu erstaunlichen Leistungen fähig: Es erkennt komplexe visuelle Muster, steuert Bewegungen und kann sich an vergangene Erlebnisse erinnern. Doch wie gelingt ihm das? Durch den Fortschritt in der künstlichen Intelligenz (KI) ist es in den vergangenen Jahren möglich geworden, dass künstliche neuronale Netzwerke ähnliche Aufgaben bewältigen – unter anderem dank leistungsstarker Software, die das effiziente Training solcher Modelle ermöglicht. Daher hoffte man, aus den Funktionsweisen der künstlichen Netze Rückschlüsse darauf ziehen zu können, wie das Gehirn tatsächlich arbeitet. Diese künstlichen Netzwerke basieren jedoch auf stark vereinfachten Neuronenmodellen, weshalb künstliche neuronale Netze bisher nur begrenzt dazu beitrugen, das Gehirn besser zu verstehen. Im Gegensatz dazu existieren auch biophysikalisch detaillierte Gehirnmodelle, welche aber nicht in der Lage sind, kognitive Aufgaben zu lösen, wie zum Beispiel visuelle Muster zu erkennen oder Erinnerungen zu speichern.
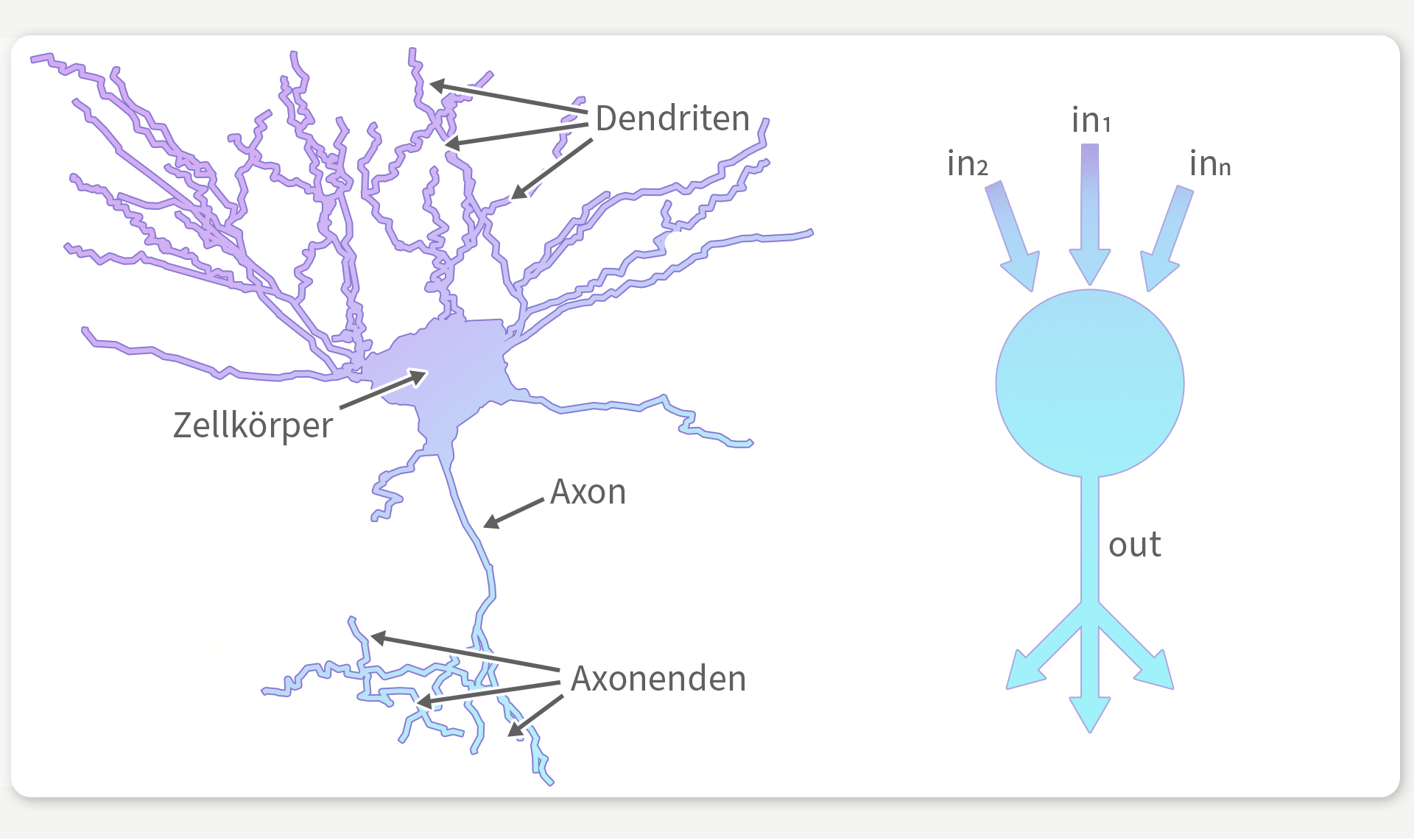
Nervenzellen im Gehirn bestehen aus einer Vielzahl von Prozessen, welche die elektrische Aktivität der Zelle beeinflussen (links). Moderne KI-Architekturen, auf denen bisherige Gehirnsimulationen oftmals beruhen, simplifizieren Neuronen stark (rechts), so dass Ergebnisse oft keine Rückschlüsse mehr auf die Funktionalität des Gehirns zulassen. © Franz-Georg Stämmele
Mit einem Team aus den Arbeitsgruppen von Jakob Macke, Philipp Berens (Universität Tübingen) und Pedro Gonçalves (VIB-Neuroelectronics Research Flanders, NERF) haben wir jetzt eine neue Software namens JAXLEY entwickelt. Die Software ermöglicht es erstmals, detaillierte Modelle des Gehirns so zu trainieren, dass sie kognitive Aufgaben lösen können. Damit schafft JAXLEY die Grundlage für eine neue Generation von Gehirnsimulationen, die tiefere Einblicke in die Funktionsweise des Gehirns und dessen Fähigkeiten erlauben werden. Die Software JAXLEY sowie Demonstrationen von Gehirnsimulationen, die sich damit realisieren lassen, wurden kürzlich in Nature Methods veröffentlicht.
Simulation von Gehirnen mit biophysikalisch detaillierten Neuronenmodellen
1963 erhielten Alan Hodgkin und Andrew Huxley den Nobelpreis für Physiologie und Medizin für ihre Beschreibung, wie zelluläre Prozesse zum „Aktionspotenzial“ führen: dem grundlegenden Signal, das von den Neuronen (fast) aller Tiere ausgesendet wird. In den folgenden Jahrzehnten erweiterten viele Neurowissenschaftler das „Hodgkin-Huxley“-Modell. Sie berücksichtigten die Morphologie einzelner Neuronen, die Beiträge Dutzender Ionen und sogar die Diffusion von Ionen innerhalb der Zellen. Um Neurowissenschaftlern die Durchführung und Simulation solcher immer komplexer werdender Simulationen zu ermöglichen, entstanden viele Toolboxen für die biophysikalische Modellierung in den Neurowissenschaften – insbesondere die NEURON-Toolbox. Der NEURON-Simulator wird seit fast 50 Jahren maintained und die Codebasis ist so groß, dass es den Anschein hat, als gäbe es kein biophysikalisches Detail, das nicht implementiert werden könnte.
Der NEURON-Simulator hat jedoch einen großen Nachteil: Da er in der Programmiersprache „C“ geschrieben ist, kann er nicht von den Möglichkeiten moderner Programmiersprachen profitieren. Moderne Deep-Learning-Bibliotheken ermöglichen das Training von Modellen mit Backpropagation, einer Methode, die misst, wie jeder Parameter zu einem Fehler beigetragen hat, und die Parameter anpasst, um zukünftige Fehler zu reduzieren. Außerdem können sie das Training mit GPUs (Grafikprozessoren, die viele Berechnungen parallel ausführen können) beschleunigen – und das praktisch ohne Aufwand für die Benutzer. Um diese Möglichkeiten für biophysikalische Simulationen des Gehirns nutzbar zu machen, haben wir einen neuen biophysikalischen Simulator entwickelt: JAXLEY (JAX & [Hodgkin-Hux]ley).
Optimierung von Gehirnsimulationen durch Backpropagation
JAXLEY ermöglicht die Simulation biophysikalischer Modelle – genau wie der NEURON-Simulator. Darüber hinaus ermöglicht es jedoch auch Backpropagation und GPU-Parallelisierung. Dadurch können biophysikalische Neuronenmodelle trainiert werden, so als wären sie künstliche neuronale Netze. JAXLEY kann die Größe der Neuronen, die Stärke der Verbindungen und die Anzahl der Ionenkanäle so trainieren, dass die Simulation mit experimentellen Messungen der neuronalen Aktivität übereinstimmt oder dass sie eine kognitive Aufgabe löst. All dies wird erreicht, weil JAXLEY vollständig in JAX geschrieben ist. JAX ist ein Deep-Learning-Framework, das sich durch die Beschleunigung langer Programme (z. B. minutenlange Gehirnsimulationen) und die Skalierung von Simulationen oder Trainings auf mehrere GPUs oder TPUs auszeichnet.
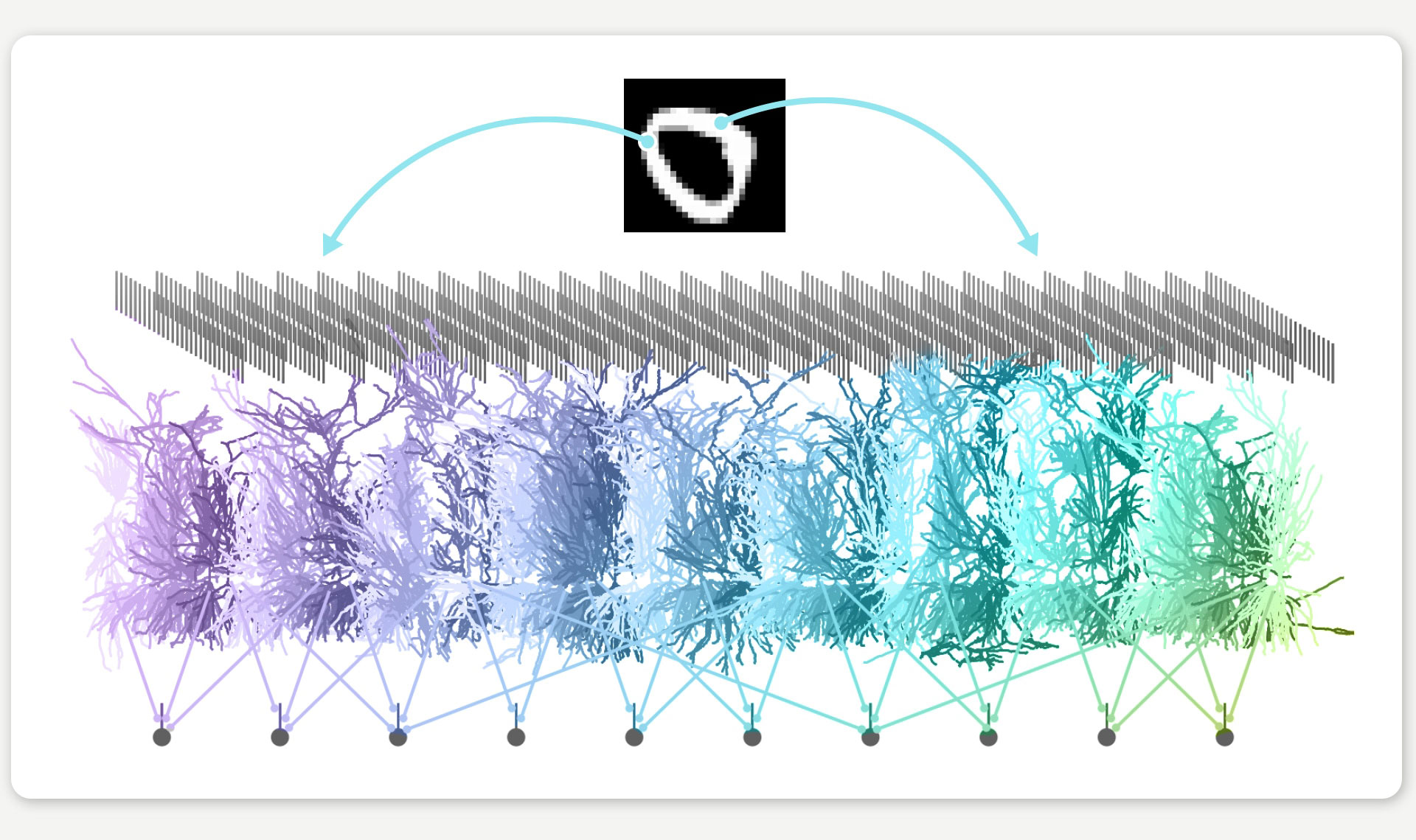
Eine Gehirnsimulation klassifiziert eine Ziffer – hier eine Null. Pixelwerte werden als Input in das Netz eingespeist (hier illustriert anhand von zwei Pixelwerten und den blauen Pfeilen) und in mehreren Layern schrittweise verarbeitet, bis am Ende die Ziffer korrekt identifiziert wird. Jedes dieser Layer besteht aus hunderten künstlichen Neuronen. JAXLEY ermöglicht es, diese künstlichen Neuronen realistisch zu simulieren und die Aufgabe gleichzeitig zu lösen. © Michael Deistler/Franz-Georg Stämmele
Mit Hilfe von JAXLEY baute unser Team Computermodelle von Teilen des Gehirns, zunächst von einzelnen Zellen. Wir trainierten die biophysikalischen Prozesse innerhalb dieser virtuellen Zellen so, dass deren Aktivität mit der Aktivität echter Gehirnzellen übereinstimmte. In einem nächsten Schritt bauten wir ein Modell der Netzhaut einer Maus, und schließlich komplexe Netzwerke, die wir mit der neuen Software so trainierten, dass sie anspruchsvolle Aufgaben ausführen konnten: Die Modelle waren dazu imstande, visuelle Muster zu erkennen oder Erinnerungen zu speichern und später wieder abzurufen.
Mit JAXLEY neuronale Mechanismen und Gehirnfunktionen untersuchen
JAXLEY macht es Neurowissenschaftlerinnen und -wissenschaftlern möglich zu untersuchen, wie neuronale Mechanismen zur Lösung von Aufgaben beitragen. Ein einzelnes Neuron ist zwar nur Mikrometer groß, enthält aber zehntausende Ionenkanäle, welche die elektrische Aktivität der Neuronen beeinflussen. JAXLEY wird es Forschenden künftig erlauben, die Komplexität des Gehirns zu erforschen und sie in Computersimulationen abzubilden. Langfristig könnten solche Simulationen auch in der Medizin Anwendung finden, etwa um neurologische Erkrankungen besser zu verstehen oder die Wirkung von Medikamenten vorab virtuell zu untersuchen.
Originalpublikation: Michael Deistler, Kyra L. Kadhim, Matthijs Pals, Jonas Beck, Ziwei Huang, Manuel Gloeckler, Janne K. Lappalainen, Cornelius Schröder, Philipp Berens, Pedro J. Gonçalves, Jakob H. Macke: Jaxley: Differentiable simulation enables large-scale training of detailed biophysical models of neural dynamics, Nature Methods, 2025. https://doi.org/10.1038/s41592-025-02895-w
Titelillustration: Franz-Georg Stämmele

Scholar Inbox: Tägliche Paper-Empfehlungen nur für dich

Kommentare